这篇文章用来记录我所有遇到的数据集它们是什么,从哪里来,什么结构,适用于哪些算法等等这些问题,具体的数据会保存在硬盘上。
以下 7 个是 SpaGCN 用到的数据,有的有图片有的没有。
# Human primary pancreatic cancer ST data (GSE111672)
下载地址
Species: Human
协议是:Spatial Transcriptomics
这里可以看到文件里面主要包含 tsv 文件、jpg 文件和 txt 文件
这个文件十分的复杂,记录一下怎么处理这上面的数据:
# 对于 tsv 文件:
这玩意是原始数据
列标题是基因名称,行标题是样本和点的标识符,数据值是表达水平
怎么读取:
1 | import pandas as pd |
绘图:
1 | import matplotlib.pyplot as plt |
# 对于 GSM3036911_PDAC-A-ST1-filtered.txt 文件
这玩意应该是提取好的数据
第一列是基因名称,标识每一行对应的基因,其余的列是采样点的表示如, 其余的和 tsv 文件类似。
完整的运行过程如下图所示:

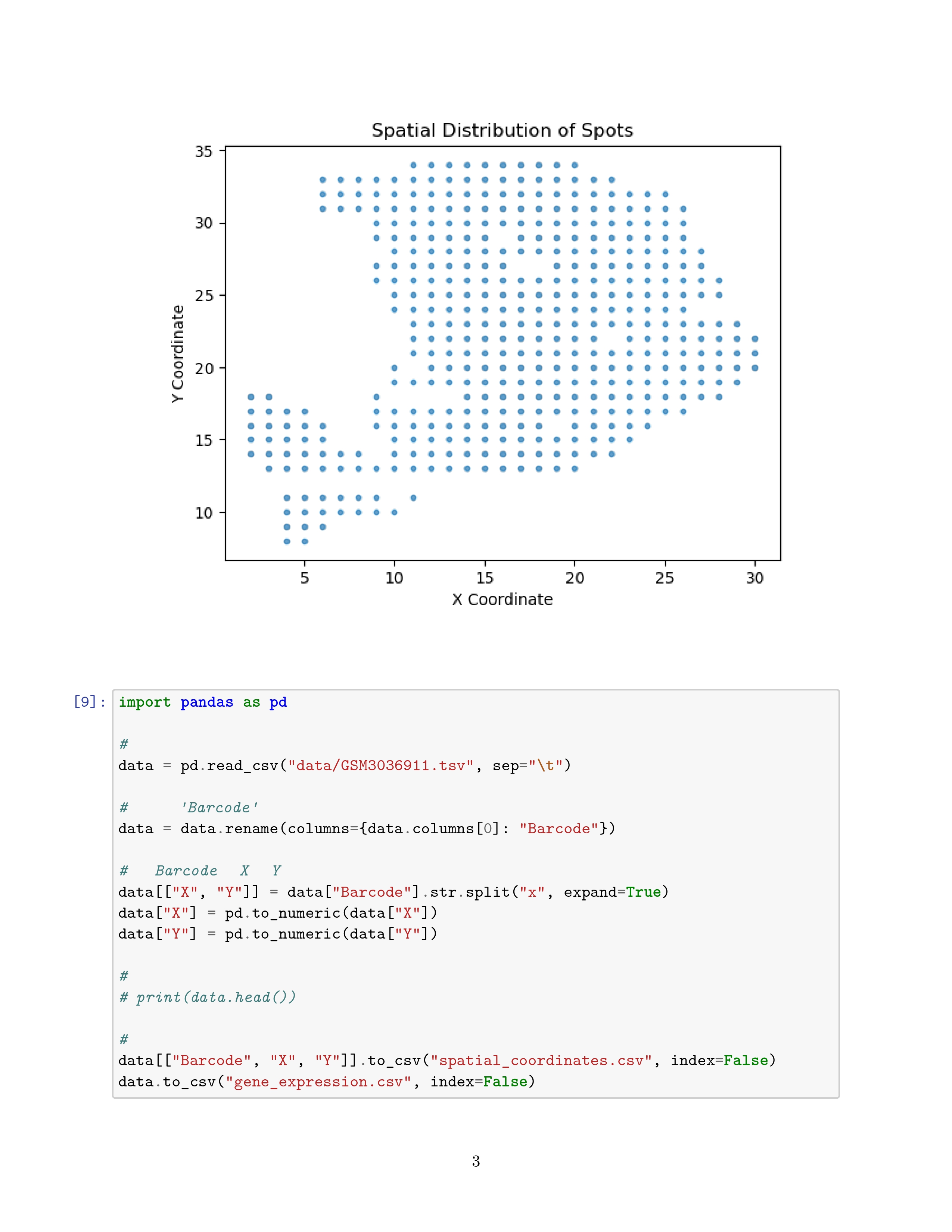

这么一看这个数据集中的 spot 数是真的少啊
# LIBD human dorsolateral prefrontal cortex, dorsolateral prefrontal cortex 10x Visium data
下载地址
Species: Human
协议是:10X Visium
保存了 h5 文件,tif 文件,position 文件
对于 151673 数据: 3639 spots. 33538 genes.
# mouse posterior brain 10x Visium data
下载地址
Species: Mouse
协议是: 10X Visium
保存了 h5 文件,tif 文件,spatial 文件夹的数据
对于数据:3353 spots. 31053 genes
# mouse cortex SLIDE-seqV2 data
下载地址
Species: Mouse
协议是: SLIDE-seqV2
保存了 h5 文件,tif 文件,position 文件
对于数据: 2560 spots. 22683 genes
# Mouse visual cortex data
下载地址
这上面的数据更加直接,在 SpaGCN 给出的数据有点抽象了。
Species: Mouse
协议是: STARmap
仅保存了一份 csv 数据
对于数据: 1207 cells. 1020 genes
# Mouse Olfactory bulb
下载地址
Species: Mouse
协议是: Spatial Transcriptomics
保存的是 csv 文件和 jpg 文件,数据直接清晰
对于数据: 262 spots. 16218 genes
# Mouse Hypothalamus data
下载地址
Species: Mouse
协议是: MERFISH
保存了 csv 文件
对于数据: 5665 cells. 161 genes.
接下来是 STAGATE 的数据集,目测没有什么好下载的,对于它的 3D 数据可以记录一下。
# Adult Mouse Brain Section 1 (Coronal)
下载地址
协议: Visium Spatial protocols
保存了 h5 文件
对于数据:
Spots detected under tissue: 2,903
Median genes per spot: 4,635
Median UMI counts per spot: 12,911
# Slide-seqV2 mouse olfactory bulb
这个和之前那个是一样的,这里点明了用的是 Puck_200127_15 的数据
spots: 20139
# Stereo-seq mouse olfactory bulb
下载地址
spots: 19109
# seven hippocampus sections profiled by Slide-seq
处理后的数据
原始数据
Puck_180531_23: spots 18509
接下来是 MENDER 的数据,挺多且挺杂的
MENDER 的数据集感觉就是在推荐它的 pysodb 的库,不用记录数据来源了,可以直接调用的
接下来是 BayesSpace,